分子细胞卓越中心张余研究组揭示了拟南芥RNA聚合酶V的转录延伸机制
文章来源:分子植物科学卓越创新中心 | 发布时间:2023-06-02 | 【打印】 【关闭】
北京时间2023年5月30日,国际学术期刊Nature Communications在线发表了中科院分子植物科学卓越创新中心/中科院合成生物学重点实验室张余研究组为“A cryo-EM structure of KTF1-bound polymerase V transcription elongation complex”的研究论文。该研究报导了包含KTF1的RNA聚合酶V(RNA polymerase V; Pol V)转录延伸复合物的高分辨率冷冻电镜结构,为理解Pol V转录延伸提供了结构基础。
陆地植物通过RNA介导的 DNA甲基化通路(RNA-directed DNA methylation pathway;RdDM)建立和部分维持基因组DNA甲基化。在该通路中,基因组DNA甲基化与Pol V的转录延伸过程紧密偶联,Pol V转录延伸过程通过两个层次的相互作用招募Argonaute4/6-siRNA复合物。第一个层次是Pol V-KTF1和 Argonaute 4/6蛋白的直接相互作用,该相互作用涉及Pol V和KTF1中包含GW基序的结构域。第二个层次是Pol V转录产生的long non-coding RNA与Argonaute4/6携带的siRNA序列匹配。随后上述复合物进一步招募DNA甲基化转移酶DRM2在Pol V的转录区域进行DNA甲基化修饰(图1)。Pol V由Pol II进化而来,但是其转录活性低于Pol II,其转录的基因组区域以及相互作用的转录调控蛋白和Pol II有显著区别。
Pol V-KTF1转录延伸复合物的冷冻电镜结构显示,Pol V具有和Pol II类似的活性中心,但是Pol V活性中心的结构单元和Pol II具有构象差异,推测该差异与Pol V的较弱转录活性相关。该研究进一步表明,Pol V具有独特的结构特征,该特征阻止其与Pol II的通用转录因子(TFIIB,TFIID,TFIIE,TFIIH,TFIIS)以及Pol IV的转录偶联因子(RDR2)相互作用,从而解释了Pol V相较Pol II以及Pol IV功能的独特性。此外,该研究揭示了KTF1的KOW5结构域和Pol V之间的相互作用,KTF1的KOW5结构域结合在Pol V的RNA退出通道附近,该结合方式使KTF1 的C端GW结构域指向Pol V转录RNA的5’末端,从而便于其招募携带siRNA的AGO4/6。该研究结果为Pol V的转录特征提供了结构解释,为进一步研究Pol V转录延伸偶联的DNA甲基化过程提供了基础(图2)。
中科院分子植物科学卓越创新中心张余组博士生章洪伟,黄坤和谷战西为论文的第一作者。该工作得到了王佳伟研究员的大力协助。本课题受到科技部重点研发计划,上海市基础研究特区计划项目资助。
论文链接:https://www.nature.com/articles/s41467-023-38619-x
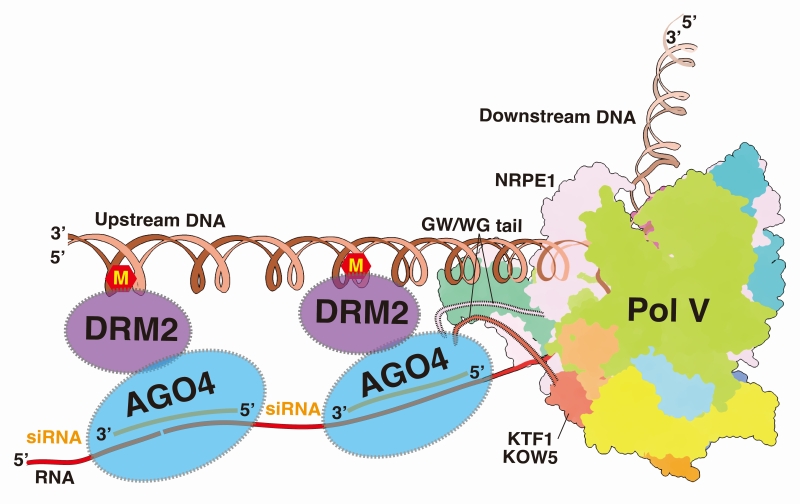
图1: Pol V转录延伸偶联DNA甲基化的模式图
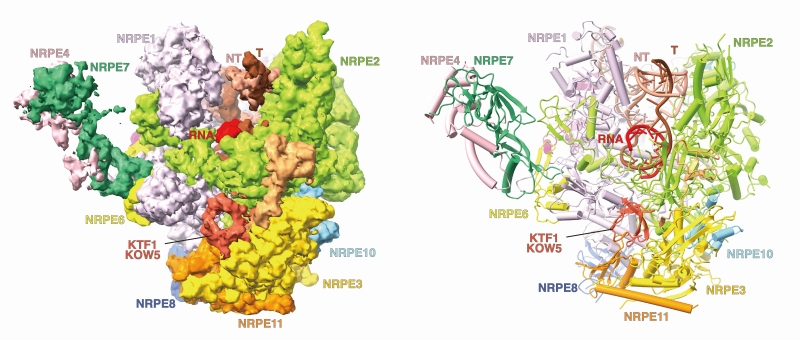
图2: Pol V-KTF1转录延伸复合物的冷冻电镜密度图和三维结构
