Nucleic Acids Res | 上海药物所开发具有用户定制功能的新型激酶谱虚拟筛选平台
文章来源:上海药物研究所 | 发布时间:2024-05-22 | 【打印】 【关闭】
2024年5月16日,中国科学院上海药物研究所郑明月课题组在Nucleic Acids Research期刊发表题为“KinomeMETA: a web platform for kinome-wide polypharmacology profiling with meta-learning”的研究论文。该研究利用结合图神经网络的元学习技术,搭建了支持用户定制的激酶多靶点活性筛选AI平台KinomeMETA(https://kinomemeta.alphama.com.cn/),为药物新型激酶靶点发现和药物筛选提供有力工具。
蛋白激酶是细胞中的关键信号传导组件,其异常激活或过表达与多种疾病相关,例如癌症、糖尿病、免疫失调和神经系统疾病等。因此,激酶靶向抑制剂是一类极具价值的治疗药物。然而,由于疾病的代偿机制和激酶结构相似性导致的脱靶效应,针对单一激酶的抑制剂往往难以持续发挥疗效并带来非预期的毒副作用,而多激酶选择性抑制剂能够在最小化用药风险的同时提供更好的疗效。为了满足更广泛的患者治疗需求,降低激酶脱靶的毒副作用风险,研究人员需要对激酶小分子探针的多重药理学效应进行系统评估。目前,激酶组范围的实验活性分析成本高昂,现有的计算工具对未充分研究的“暗”激酶及激酶突变体缺乏预测能力。因此,需要开发具有更广泛应用场景的AI算法,通过充分利用内部或自有实验数据,大规模筛选潜在的激酶靶点和多靶点选择性激酶小分子探针。
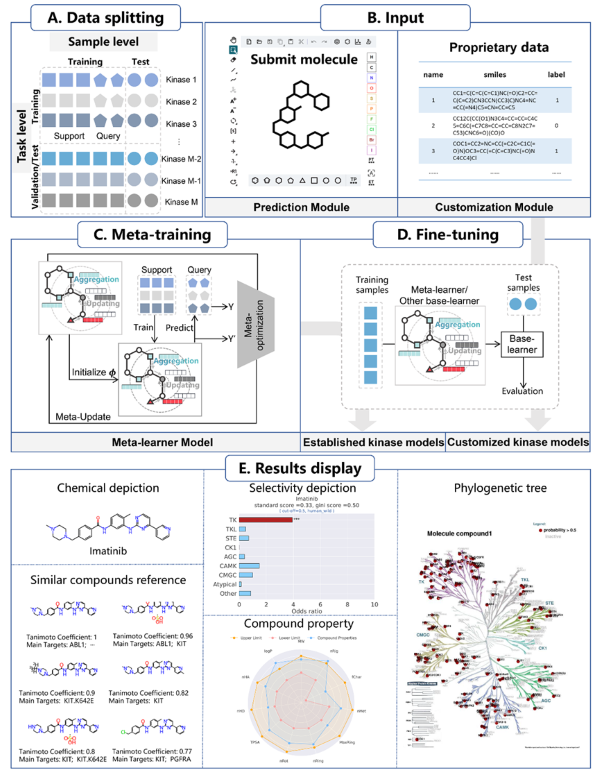
图1. KinomeMETA平台构建与使用流程概览
在该项研究中,研究人员基于前期开发的分子特征提取图神经网络AttentiveFP架构,利用元学习方法Reptile的快速适应能力,从超过61万个激酶活性数据中学习不同激酶中活性化合物的作用模式。KinomeMETA平台通过元训练和微调过程构建,包含了“定制”和“预测”两大核心模块。其中,“定制”模块是激酶活性预测领域中的全新功能,该功能允许用户使用私有数据构建全新激酶模型或增强已有激酶模型的预测能力,打破了计算/实验领域之间的研究壁垒。KinomMETA可应用范围广,其“预测”模块可预测化合物对661个野生激酶及临床相关激酶突变体的抑制活性概率。该平台还提供了化合物激酶选择性分析、分子性质评估及相似抑制剂鉴别三种结果分析功能,辅助激酶药物研究人员开展进一步研究。
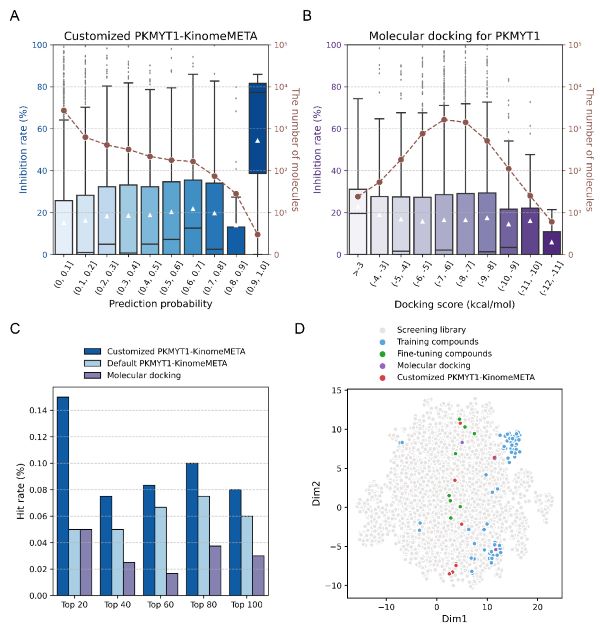
图2.使用KinomeMETA平台对内部化合物库进行PKMYT1小分子探针的虚拟筛选效率评估
此外,研究人员利用KinomeMETA前期对“暗”激酶PKMYT1小分子探针筛选的研究结果,基于KinomeMETA平台的“定制”功能增强模型,筛选了实验室内部的化合物,并进行了实验验证。结果表明,KinomeMETA平台通过少数活性化合物迅速增强了预测能力,对PKMYT1活性小分子探针的发现效率始终优于分子对接方法。
KinomeMETA平台提供了预测激酶组范围活性的“预测”功能和高效利用私有数据的“定制”功能,通过迭代的干/湿实验循环,可以有效应对药物发现中的低数据场景,显著加速新型激酶小分子探针和可成药激酶靶点的发现。
德州学院李召军博士、中国科学院上海药物研究所研究生曲宁和上海科技大学研究生周敬怡为论文的共同第一作者。中国科学院上海药物研究所药物发现与设计中心(DDDC)李叙潼副研究员、郑明月研究员与张素林副研究员为论文通讯作者。本研究得到了国家自然科学基金、国家重点研发专项、中国博士后科学基金、中国科学院青年创新促进会、上海药物研究所与上海中医药大学中医药创新团队联合研究项目、以及上海市科技重大专项资助。
原文链接:Li. et al., KinomeMETA: a web platform for kinome-wide polypharmacology profiling with meta-learning, Nucleic Acids Research (2024), https://doi.org/10.1093/nar/gkae380.
