分子植物科学卓越创新中心张余研究组揭示植物Pol IV转录延伸倒退机制
文章来源:分子植物科学卓越创新中心 | 发布时间:2024-08-26 | 【打印】 【关闭】
DNA甲基化修饰对于维持植物基因组稳定性和调控基因表达至关重要。RNA 聚合酶Pol IV和RDR2是植物RNA-directed DNA methylation通路的重要组成成员。中国科学院分子植物科学卓越创新中心张余团队前期研究发现Pol IV和RDR2形成一个双RNA聚合酶复合物,并提出了 Pol IV-RDR2复合物以双链DNA为模板合成双链RNA的“RNA内部传递”的模型(PMID: 34941388)。在上述模型中,研究人员提出Pol IV首先在基因组DNA上前进合成一定长度的RNA,随后其发生倒退将RNA从其催化中心传递到RDR2催化中心合成双链RNA。该模型的关键步骤是Pol IV在转录延伸的过程中发生转录倒退,从而触发Pol IV RNA从内部通道进入RDR2的催化中心。但Pol IV为什么能够发生高效的转录倒退?这一关键的科学问题目前尚未阐明。
2024年8月23日,中国科学院分子植物科学卓越创新中心张余团队在Science Advances上发表题为“Transcription elongation of the plant RNA polymerase IV is prone to backtracking”的研究论文,研究结果揭示了Pol IV在从Pol II进化的过程中,其催化中心发生变化从而赋予其倾向转录倒退的特征,进一步支持了Pol IV-RDR2复合物独特的“RNA内部传递”工作模型。
在RNA聚合酶往RNA3’端添加核苷酸延长RNA链的过程中,RNA聚合酶催化中心结合NTP,随后在其关键结构元件的帮助下(catalytic loop, bridge helix和trigger loop)催化磷酸二酯键形成、释放焦磷酸、催化中心移位空出NTP结合位点。整个过程叫做核苷酸添加(Nucleotide-addition cycle;NAC)。研究人员解析了Pol IV 的NAC过程中四个不同状态的三维结构。通过和Pol II的类似结构对比,研究人员发现Pol IV在催化 NTP 的过程中其催化中心结构元件发生不同于Pol II的构象变化,从而导致Pol IV具有较弱的NTP结合能力、较低的催化活性以及较弱的解旋下游DNA活性。综上这些特征提示Pol IV转录延伸的过程中具有较弱的往前行进能力,倾向于在遇到障碍时发生转录倒退。
RNA聚合酶在转录延伸过程中一旦发生转录倒退,RNA的3’端会占据NTP结合通道,从而抑制转录延伸。细菌RNAP,古菌RNAP以及高等动植物的Pol I, Pol II和Pol III 其通过自身的水解活性或者转录校正因子水解backtracked RNA。研究人员通过生化实验证明Pol IV转录延伸过程中backtracked RNA能够很好的被保护不被降解。研究人员发现Pol IV具有极低的水解RNA活性,通过解析Pol IV backtracked state的复合物结构,发现Pol IV催化中心的结构变化使其不能够固定backtracked RNA发生水解反应。其次,通过进一步生化实验和结构证据表明Pol II的转录校正因子不能作用Pol IV 切割backtracked RNA。以上机制确保Pol IV backtracked RNA能够完整进入到RDR2催化活性中心,保证双链RNA的高效合成。
中国科学院分子植物科学卓越创新中心张余研究组已毕业博士研究生方城力和黄坤为该论文共同第一作者,张余研究员为通讯作者。本研究受到科技部重点研发计划、国家自然科学基金面上项目、上海市基础研究特区项目资助。
论文链接:https://www.science.org/doi/10.1126/sciadv.adq3087
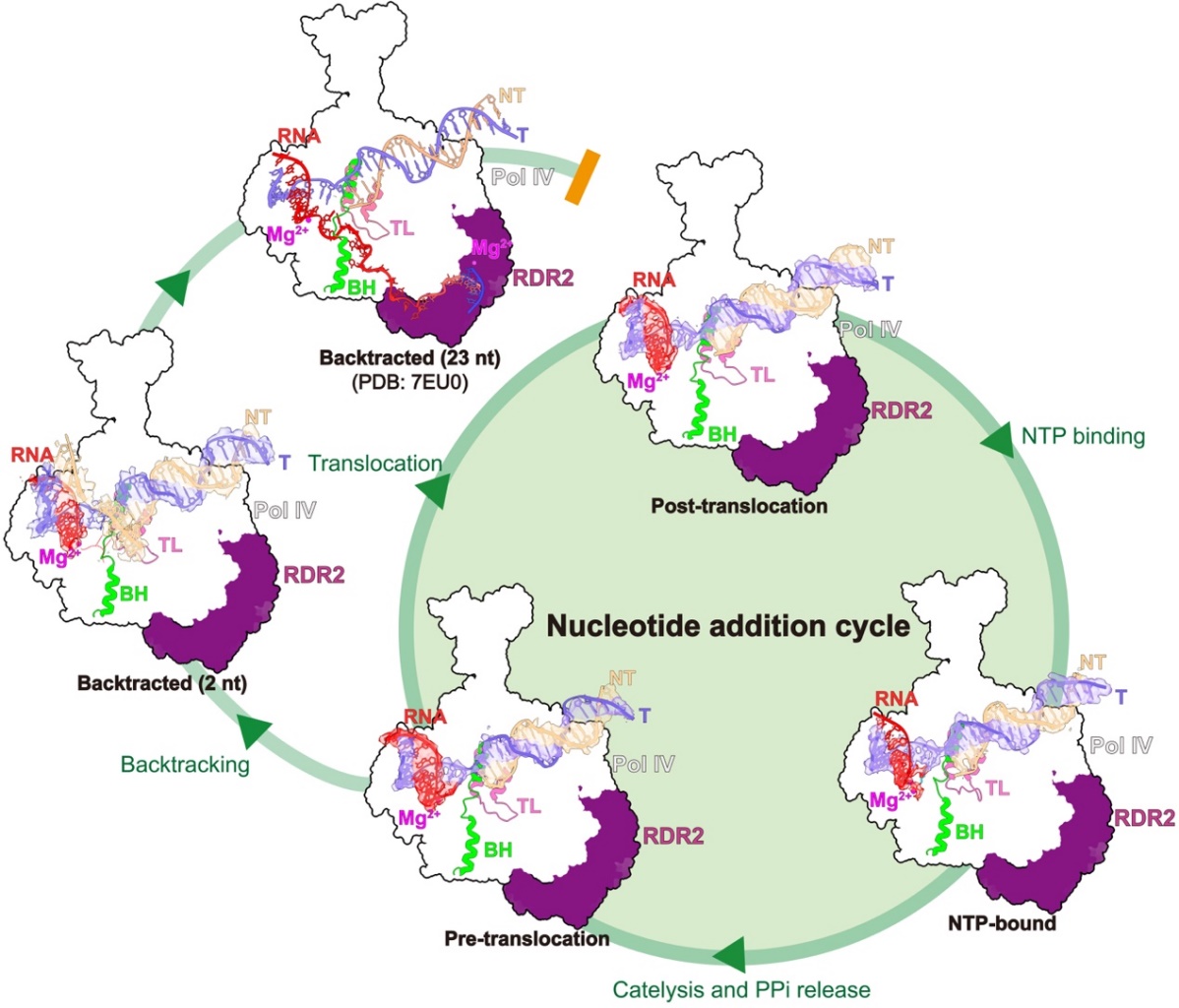
Pol IV-RDR2的核苷酸添加循环示意图
