营养与健康所邵振与詹丽杏研究组合作开发蛋白质组比较算法解析红细胞分化和肝癌异质性
文章来源:上海营养与健康研究所 | 发布时间:2024-10-17 | 【打印】 【关闭】
10月14日,国际期刊Genome Biology在线发表了中国科学院上海营养与健康研究所邵振研究组、詹丽杏研究组与中国科学院分子细胞科学卓越创新中心柳欣研究组题为”zMAP toolset: model‑based analysis of large‑scale proteomic data via a variance stabilizing z‑transformation”的合作研究成果。该研究针对当前TMT和iTRAQ等定量蛋白质组数据缺乏多样本比较分析工具的瓶颈,开发了zMAP工具集,并应用它解析了红细胞分化过程和肝癌病人组织之间的蛋白质组动态变化。
TMT和iTRAQ等重同位素标记技术已广泛用于蛋白质组定量分析,为研究组织发育和疾病进程中的蛋白质表达变化提供了基础。现有的蛋白质组差异分析工具主要面向两个或两组样本之间比较,能进行多样本比较的工具有限。尤其是这些平台生成的蛋白质组数据往往包含强烈的批次效应,导致能够整合多个质谱实验批次数据开展定量比较分析的工具更是匮乏,局限了它们在大规模生物样本分析中的应用。针对该瓶颈,研究者开发了zMAP和reverse-zMAP两个新计算模型,并基于它们搭建了一个多功能的蛋白质组数据分析平台,面向基于不同实验设计的多批次多样本数据开展统计比较和下游整合分析(图1)。
研究人员应用zMAP分析了一个已发表的红细胞分化蛋白质组数据集,包含4种细胞类型(成年阶段造血干祖细胞A0及体外分化5天后的原红细胞A5;胚胎阶段造血干祖细胞F0及分化5天后的原红细胞F5)和5次4通道iTRAQ实验生成的5组生物学重复。zMAP识别了2290个显著高变化蛋白质,并基于z统计值对它们的表达模式聚类,发现很多仅在胚胎红细胞分化过程中上调的蛋白质。研究人员从中选择了S100A8、S100A9、CHI3L1和PTEN,验证了这些基因在相应分化过程中发挥了不可或缺的作用。
此外,研究人员还应用reverse-zMAP分析了一个包含159例原发肝癌患者肿瘤和瘤旁正常对照组织的公开TMT蛋白质组数据集,鉴定出3097个高变蛋白质。基于它们z统计值矩阵的主成分分析可以将两类样本清楚分开。结合z统计值和病人生存数据的Cox比例风险回归分析鉴定了大量预后相关蛋白质。它们中69.6%与第一主成分(PC1)显著相关,即在肿瘤和正常对照组织之间差异表达,其中很多如NPC1和UBE2C可作为肝癌进展分子标志物。研究人员基于PC1得分对肿瘤样本进行层次聚类,将它们分为三个亚组,发现该分型能够比常用的TNM和BCLC分期更好地解释病人预后差异;另一方面,基于z统计值的基因集变异分析(GSVA)指出,PC1得分较高的癌旁对照肝组织样本表现出肝代谢通路下调和免疫相关通路上调这一特殊状态,可能为肝癌发生“炎癌转换”过程蛋白质组的转变模式提供有价值线索。上述两方面发现表明这两类病人组织样本沿PC1分布的异质性均具有可观的生物学意义。研究人员还整合z统计值和肿瘤体细胞突变数据开展了蛋白质表达数量性状位点分析(pQTL)。5个高频突变基因中,携带TSC2突变的肿瘤样本PC1得分较高,并且患者预后较差,其肿瘤中TSC2蛋白质表达显著降低。根据pQTL结果,发现TSC2突变下调的均为对病人生存有利的蛋白质,而上调的均为对病人生存不利的蛋白质。最后,研究人员验证了NPC1和UBE2C对肝癌细胞增殖和侵袭的促进作用以及TSC2的抑癌作用。
中国科学院上海营养与健康研究所博士后桂秀琪、研究生黄静、吴燕君、郭烜和中国科学院分子细胞科学卓越创新中心研究生阮林杰为该论文共同第一作者。中国科学院上海营养与健康研究所邵振研究员、涂世奇副研究员、詹丽杏研究员和中国科学院分子细胞科学卓越创新中心柳欣研究员为该论文共同通讯作者。该工作得到国家自然科学基金委、科技部、中国科学院等多项基金支持。研究使用的所有组学数据均为已公开发表数据。
原文链接::https://genomebiology.biomedcentral.com/articles/10.1186/s13059-024-03382-9
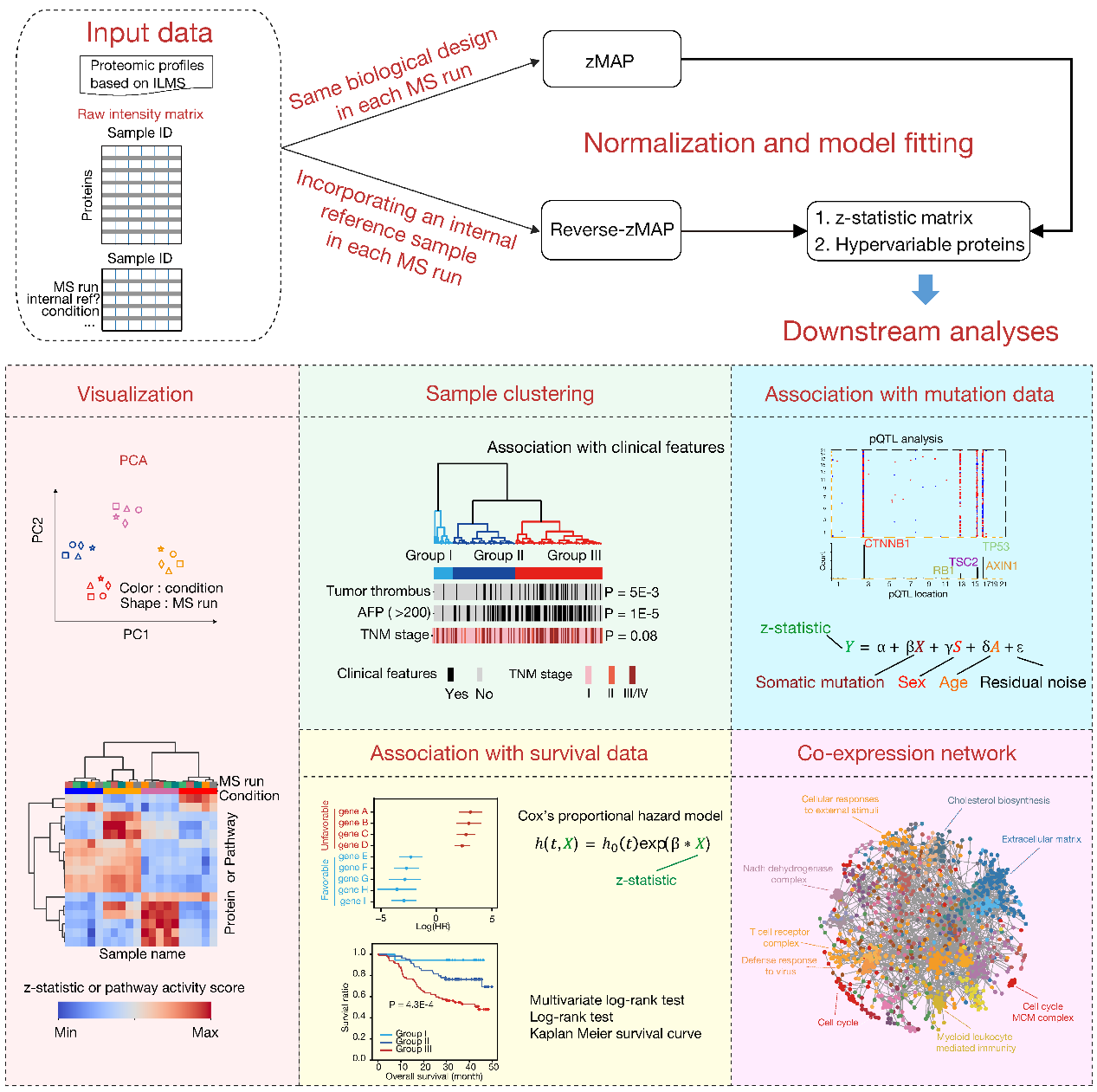
图1:蛋白质组数据分析平台zMAP的核心架构
